
总体来看是弱监督学习的领域,没有接触过,主要需要学习的是两个算法和如何使用深度学习优化其中的参数:
- 多实例学习 MIL
- 正-无标注学习 PUlearning
论文背景和假设
这篇论文需要一定的背景
假设现在提供一个病理切片图像,其中只有部分的区域是癌变而其余部分是正常的。
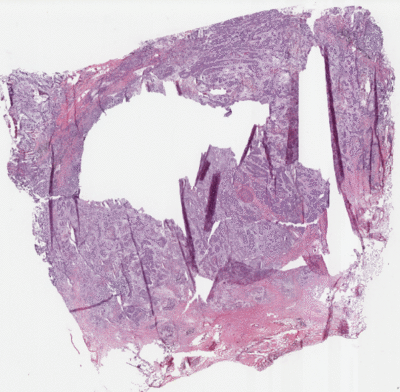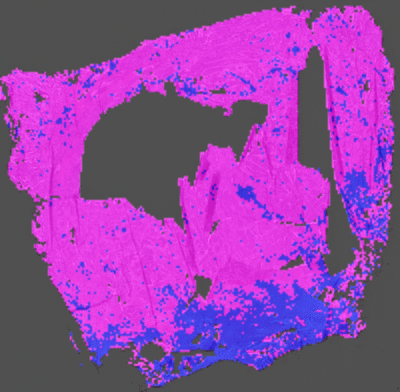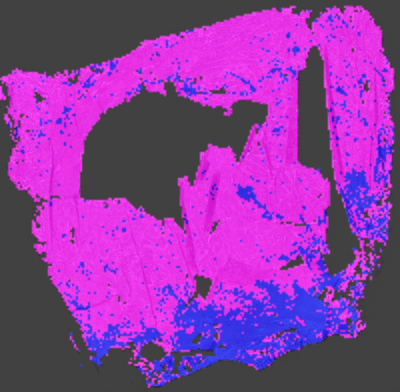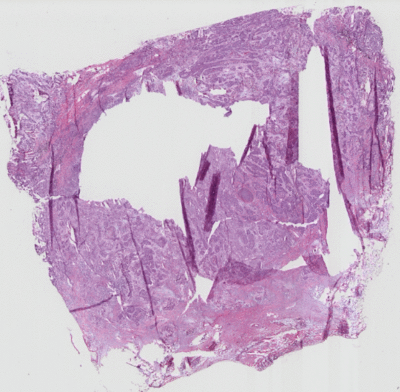
但是实际上所提供的mask大多是这样的:
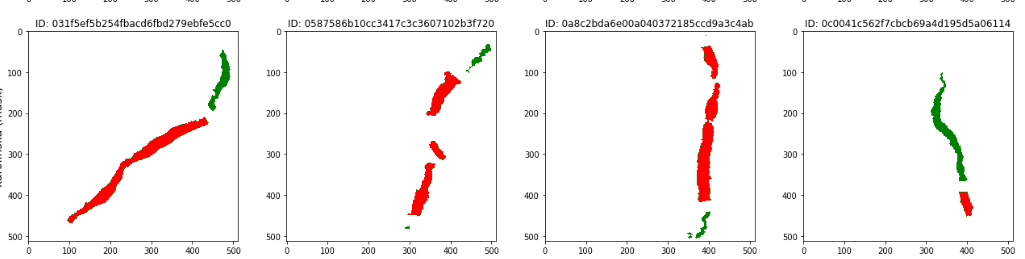
所以很大程度上无法分割出具体是哪些点出了问题,即无法实现像素级别的识别,也就引出了这个论文要求。
多实例学习(MIL)
找到了一个非常易于理解的解释,以MNIST数据集为例,里面有0~9个标签表示对应的数字。每次从MNIST数据集中取n个数据(差不多3~10)放到一个袋子里。其中我们把包含了1的袋子设置标签为1,其余的为0。此时,如果忽略掉数据只考虑袋子,就得到了两种类别的袋子0、1。在这个例子下,多实例学习的目标就是,已知袋子的标签,需要知道是袋子里哪个数据决定了这个标签。
那么对于现在这个病理图像分割,我们已知类别,所以需要找到一整张切片中导致其为某个类别的区域或特征,因此该课题可以使用多实例学习解决。具体的repo:
https://github.com/MarvinLer/tcga_segmentation
正-无标注学习(PUL)
正无标注学习的场景是只有正标注的数据和未标注的数据,在此,我们可以视病理切片掩码中,绿色的一定是有问题的(正标注),红色是可能有问题的(无标注),由此就满足了PUL的条件。基于PUL的分割算法:

https://github.com/RxstydnR/PUlearning_segmentation
数据集使用
这里使用的是Prostate cANcer graDe Assessment (PANDA数据集),前列腺等级分类挑战中的数据。其主要提供了:
- 组织切片图片
- 癌变区域掩码图片
- isup类别和格里森等级
其中对实验有效的是组织切片图片和癌变区域掩码图片。数据集大小为411G。
存在问题
效果可能难以进行评估,目前只能通过与其他方案进行对比以凸显效果
数据量大且有很多低质量数据,数据处理步骤重要而且可能会影响最终结果
数据下载
使用kaggle页面进行下载(因为使用API下载过于频繁被封号了)
数据处理
数据分析部分代码 main.ipynb
#每张tiff包含三层,取第一层为最清晰的
#显示掩码
def display_masks(slides):
f, ax = plt.subplots(5,3, figsize=(18,22))
for i, slide in enumerate(slides):
mask = openslide.OpenSlide(os.path.join(mask_dir, f'{slide}_mask.tiff'))
mask_data = mask.read_region((0,0), mask.level_count - 1, mask.level_dimensions[-1])
cmap = matplotlib.colors.ListedColormap(['black', 'gray', 'green', 'yellow', 'orange', 'red'])
ax[i//3, i%3].imshow(np.asarray(mask_data)[:,:,0], cmap=cmap, interpolation='nearest', vmin=0, vmax=5)
mask.close()
ax[i//3, i%3].axis('off')
image_id = slide
data_provider = train.loc[slide, 'data_provider']
isup_grade = train.loc[slide, 'isup_grade']
gleason_score = train.loc[slide, 'gleason_score']
ax[i//3, i%3].set_title(f"ID: {image_id}\nSource: {data_provider} ISUP: {isup_grade} Gleason: {gleason_score}")
f.tight_layout()
plt.show()
slides = os.listdir(origin_dir)[40:55]
slides_id = [i.split(".")[0] for i in slides]
display_masks(slides_id)

对于mask图片同样有三层,每层大概是H*W*1C即通道数为1,对于每个像素点的值,有三种0、1、2,显示出来为黑色、灰色、绿色。其中黑色表示背景、灰色为正常部位、绿色为异常部位
由于背景内容较大,在提取时,当图片中1和2的像素点大于0时进行分割,分割得到的尺寸为512*512
import gc
def tile_single(slide):
image = openslide.OpenSlide(os.path.join(origin_dir, f'{slide}.tiff'))
mask = openslide.OpenSlide(os.path.join(mask_dir, f'{slide}_mask.tiff'))
width, height = image.level_dimensions[0]
# mask_data = np.asarray(mask.read_region((0,0), 0, mask.level_dimensions[0])).copy()
# image_data = np.asarray(image.read_region((0,0), 0, image.level_dimensions[0])).copy()
for i in range(0,width-512,512):
for j in range(0, height-512,512):
mask_img_tile = np.asarray(mask.read_region((i,j), 0, (512,512))).copy()
mask_img_tile = np.reshape(mask_img_tile[:,:,0],(512,512,1))
count = pd.value_counts((mask_img_tile).ravel())
try:
nums_zero = count[0]
if(count[0] < 196608):
origin_img_tile = np.asarray(image.read_region((i,j), 0, (512,512))).copy()
origin_img_tile = cv2.cvtColor(origin_img_tile, cv2.COLOR_RGBA2RGB)
mask_img_tile[mask_img_tile == 1] = 127
mask_img_tile[mask_img_tile == 2] = 255
cv2.imwrite("temp/{}_{}_{}.png".format(slide,i,j),origin_img_tile)
cv2.imwrite("temp/{}_{}_{}_mask.png".format(slide,i,j),mask_img_tile)
except KeyError:
origin_img_tile = np.asarray(image.read_region((i,j), 0, (512,512))).copy()
origin_img_tile = cv2.cvtColor(origin_img_tile, cv2.COLOR_RGBA2RGB)
mask_img_tile[mask_img_tile == 1] = 127
mask_img_tile[mask_img_tile == 2] = 255
cv2.imwrite("temp/{}_{}_{}.png".format(slide,i,j),origin_img_tile)
cv2.imwrite("temp/{}_{}_{}_mask.png".format(slide,i,j),mask_img_tile)
gc.collect()
slide = os.listdir(origin_dir)[40].split(".")[0]
slide = "001c62abd11fa4b57bf7a6c603a11bb9"
print(slide)
tile_single(slide)
karo_3 = train[train["isup_grade"] >= 3][train["data_provider"] == "karolinska"].index
for i in karo_3[:200]:
tile_single(i)
进度
阅读了相关论文和代码并进行了一定的可行性分析。目前主要在进行数据的下载和MIL模型的测试。
进度
走通了数据集处理流程和模型训练流程。
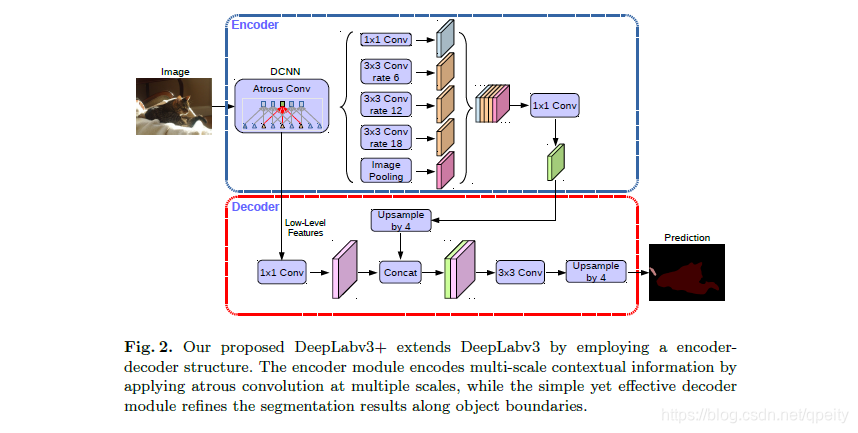
DeepLabv3+
用于分割的模型,在一些数据集上达到了sota效果
数据集加载
import os
import numpy as np
from PIL import Image
import torch.utils.data as data
class TCGASegmentation(data.Dataset):
"""TCGA Segmentation Dataset.
Args:
root (string): Root directory of the TCGA Dataset.
image_set (string, optional): Select the image_set to use, `train` or `val`
transform (callable, optional): A function/transform that takes in an PIL image
and returns a transformed version. E.g, `transforms.RandomCrop`
"""
def __init__(self, root, image_set='train', transform=None):
self.root = os.path.expanduser(root)
self.transform = transform
self.image_set = image_set
image_dir = os.path.join(self.root, 'img_dir', image_set)
mask_dir = os.path.join(self.root, 'ann_dir', image_set)
if not os.path.isdir(image_dir) or not os.path.isdir(mask_dir):
raise RuntimeError('Dataset not found or corrupted. Please check your dataset directory.')
self.images = sorted([os.path.join(image_dir, img) for img in os.listdir(image_dir) if img.endswith('.png')])
self.masks = sorted([os.path.join(mask_dir, mask) for mask in os.listdir(mask_dir) if mask.endswith('_mask.png')])
assert (len(self.images) == len(self.masks))
def __getitem__(self, index):
"""
Args:
index (int): Index
Returns:
tuple: (image, target) where target is the image segmentation.
"""
img = Image.open(self.images[index]).convert('RGB')
target = Image.open(self.masks[index])
if self.transform is not None:
img, target = self.transform(img, target)
return img, target
def __len__(self):
return len(self.images)
@staticmethod
def decode_target(mask):
"""decode semantic mask to RGB image"""
label_colors = np.array([
[0, 0, 0], # Unlabeled
[128, 128, 128], # Background
[255, 255, 255] # Positive
])
return label_colors[mask]

PUlearning实践
PUlearning在某种程度上也可以看作是两次迭代的训练,但是问题是容易成为强监督学习过拟合,如何控制是一个问题

目前有了一个新的思路,取一个正确标注的数据集,通过使用图像处理等操作,扩散负标签区域从而使任务符合PUlearning,并通过训练得到的结果与正确的结果进行比对从而确认上述思路是否正确。
关于代码的运行
训练
python main.py --data_root ../TCGA --dataset tcga --enable_vis --vis_port 12345 --continue_training --model "deeplabv3plus_xception"
推理
python predict.py --input ../TCGA/img_dir/train --dataset tcga --model "deeplabv3plus_xception" --save_val_results_to ../TCGA/ann_dir/one_cur/ --ckpt ./checkpoints/best_deeplabv3plus_xception_tcga_os16.pth
中期报告之后的进展
之前的模型效果确实不好, 但是大致也找到了原因:语义分割模型和医学影像分割模型的差距还是相当大的,因此决定使用医学影像分割模型——TransU-Net,使用了Transformer的U-Net,一方面是使用了Transformer结构的模型会有较好的效果,但是同时没有使用一些更为先进的模型来防止在弱监督时过拟合,
在使用了TransU-Net并做了强监督学习的测试,发现效果确实相当好,miou大概能到80%(该数值在绝大多数医学影像分割任务已经可以达到SOTA效果了),因此,使用该模型的思路是正确的。由于原作者的代码过于恶心,因此对其进行了大规模的重构和修改并增加了大量的功能。经过一段时间的调参和测试之后,目前在弱监督的数据集训练上已经可以获得均衡的正负样本区域,但是仍然存在一些较大的问题,目前有如下改进思路:做正则化,每次部分的从中获取数据用作下一轮的测试,使用计算机视觉的方案做数据处理以达到更好的效果。
弱监督的训练
python .\main.py --model_path weakly.pth --mode train --train_mode weakly --train_path temp --test_path temp
#其中的--train_path 和--test_path只是参数需要
强监督的训练
python .\main.py --model_path model.pth --mode train --train_path ../TCGA/dataForU/train --test_path ../TCGA/dataForU/val --train_mode default
